これまでの主な研究内容
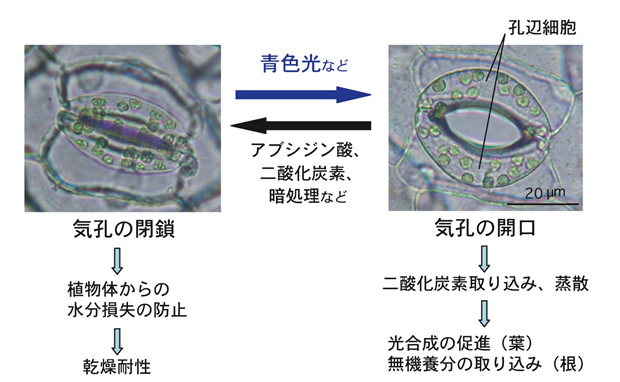
陸生植物の表皮に存在する気孔は、一対の孔辺細胞に囲まれた孔で、変転する環境に応じて開閉し、光合成に必要な二酸化炭素の取り込みや蒸散など植物と大気間のガス交換を調節している。孔辺細胞は、光合成が盛んに行われる光照射下、特にシグナルとして作用する青色光に反応して気孔を開き、ガス交換を促進し、乾燥ストレスに曝されると植物ホルモン・アブシジン酸に応答して気孔を閉じ、植物体からの水分損失を防ぐ。気孔の開閉は、孔辺細胞が外部からのシグナルに的確に応答してシグナル伝達を行い、各種イオンの流入・流出に関わるポンプやチャネル活性を調節し、最終的に孔辺細胞の体積が変動することにより引き起こされる高次の生命現象である(下図)。
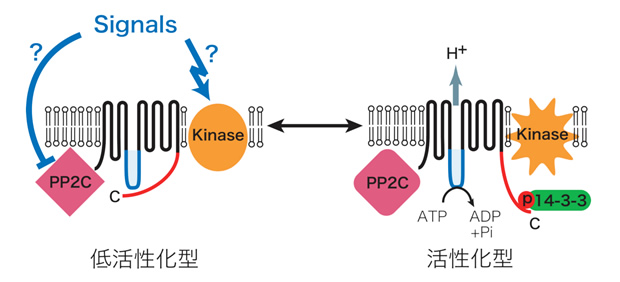
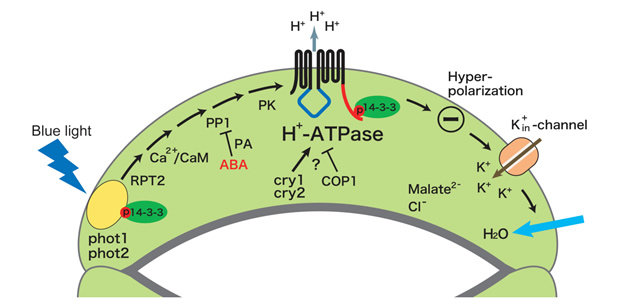
青色光は、孔辺細胞のATP要求性細胞膜H+ポンプを活性化し、細胞膜の過分極を引き起こす。これに応答して電位依存性K+チャネルが孔辺細胞内へ大量のK+を流入させ、それに伴い水が取り込まれ気孔開口に至る(1)。しかしながら、青色光受容体やH+ポンプの実体など、シグナル伝達の分子レベルでの解明はほとんど進んでいなかった。そこで、まず、青色光に活性化されるH+ポンプについて、ソラマメ孔辺細胞プロトプラストにおけるATPase活性測定法を確立して解析を行い、その実体が植物特有のP型ATPaseの細胞膜H+-ATPaseであることを明らかにした。また、in vivo [32P]ラベルした孔辺細胞を用いた解析により、青色光はH+-ATPaseのC末端領域のリン酸化を引き起こし、リン酸化されたC末端に14-3-3 蛋白質が結合すること見出した(2)。さらに、活性化の分子機構について解析を進め、定常状態で自己阻害的に働いているC末端領域のThr950が青色光によりリン酸化され、そこに14-3-3蛋白質が結合することによってH+-ATPaseが活性化されることを証明した(3)。細胞膜H+-ATPaseは、気孔の開口だけでなく、植物細胞おける二次輸送体を介した物質輸送や膜電位・細胞内外pHの調節など極めて重要な役割を果たしているが、細胞内での活性調節機構は不明であった。本成果は、生理的シグナルによる細胞膜H+-ATPaseの活性化機構を示した初めての例となった(4)(下図)。
この研究過程において、ソラマメ孔辺細胞において青色光により自己リン酸化する125 kDa蛋白質を発見し、その蛋白質が芽生えの光屈性の青色光受容体として同定されていたフォトトロピンであることを突き止めた(5)。そこで、モデル植物シロイヌナズナのフォトトロピン変異体(phot1, phot2)における気孔の青色光反応を調べ、phot1とphot2の各シングル変異体では正常だが、2重変異体においては気孔の青色光反応が全く見られないことを示し、phot1とphot2が重複して細胞膜H+-ATPase活性化・気孔開口の青色光受容体として機能していることを証明した(6, 7)(下図)。
フォトトロピンは、Ser/Thrキナーゼドメインを持つ光受容体であり、気孔開口のみならず、光屈性、葉緑体光定位運動や葉の横伸展の光受容体として機能する。そこで、フォトトロピンにより引き起こされる諸反応の生理的役割について解析を行い、フォトトロピンは、弱い光環境のもとで上記の諸反応を引き起こして光合成能を増大させ、結果的に植物の生長を促進していることを実証した(8)。
以上の研究と並行して、ペンシルバニア州立大学のAssmann教授らとの共同研究により、アブシジン酸による気孔閉鎖に関与するプロテインキナーゼ AAPKの細胞内基質の探索を行い、アブシジン酸に応答してAAPKがヘテロ核RNA結合蛋白質A/B(AKIP1)をリン酸化することを示した(9)。さらに、孔辺細胞の細胞膜H+-ATPaseが生理的濃度のCa2+により可逆的に阻害されることを見出し、アブシジン酸による孔辺細胞細胞質のCa2+濃度上昇に伴い細胞膜H+-ATPaseが阻害され、気孔閉鎖が促進されている可能性を示した(10)。
また、2003年10月から1年間滞在したSalk生物学研究所のJ. Chory教授の研究室では、植物の正常な成長や分化において重要な役割を果たす植物ホルモン・ブラシノステロイド(BR)の受容機構について研究を行い、受容体型キナーゼBRI1がBR受容体であることを証明した(11)。
今後の研究目的
これまでの研究の主な成果として、気孔開口に関わる青色光受容体(フォトトロピン)や効果器(細胞膜H+-ATPase)の実体を明らかにしたが、フォトトロピンからH+-ATPase活性化に至るシグナル伝達やH+-ATPaseのリン酸化反応を触媒する酵素は未解明である。現在は、気孔の開閉シグナル伝達機構やH+-ATPaseの活性調節の分子機構の全容解明を目指し、生理・生化学・分子遺伝学的手法を駆使して、以下のような研究進めている。
気孔開閉の分子機構についての生理・生化学的解析
モデル植物シロイヌナズナやソラマメから単離した高純度孔辺細胞プロトプラストを用い、細胞膜H+-ATPase複合体解析や青色光やアブシジン酸に応答した蛋白質リン酸化反応の解析、孔辺細胞特異的に発現する遺伝子の機能解析を進めている。
分子遺伝学的手法による気孔開度変異体の単離
植物の気孔開度測定は大変時間を要するため、ハイスループットな選抜が可能な様々な方法によりスクリーニングを行い、シロイヌナズナにおける新奇の気孔開度変異体の単離と原因遺伝子の機能解析を進めている。
細胞膜H+-ATPaseの活性調節機構と突然変異体の単離
気孔孔辺細胞を用いた研究により明らかとなったC末端のリン酸化と14-3-3蛋白質の結合によるH+-ATPaseの活性化機構は、孔辺細胞以外の細胞でも見られる生理的な活性化機構であることが証明されつつある。しかしながら、C末端リン酸化に関わるプロテインキナーゼやホスファターゼの実体は明らかとなっていない。そこで、生化学的手法によるC末端のリン酸化の解析(12)、さらに、シロイヌナズナを用いた様々なスクリーニングにより、新奇の細胞膜H+-ATPaseの活性調節変異体の単離を進めている。
参考文献
- Shimazaki K, Doi M. Assmann SM, Kinoshita T. (2007) Light regulation of stomatal movement. Annu. Rev. Plant Biol. 58:219-247.
- Kinoshita T, Shimazaki K. (1999) Blue light activates the plasma membrane H+-ATPase by phosphorylation of the C-terminus in stomatal guard cells. EMBO J. 18: 5548-5558.
- Kinoshita T, Shimazaki K. (2002) Biochemical evidence for the requirement of 14-3-3 protein binding in activation of the guard-cell plasma membrane H+-ATPase by blue light. Plant Cell Physiol. 43:1359-1365.
- 木下俊則、島崎研一郎「気孔の開口を駆動する細胞膜H+-ATPase」蛋白質核酸酵素 51, 871-876. (2006年)
- Kinoshita T, Emi T, Tomonaga M, Sakamoto K, Shigenaga A, Doi M, Shimazaki K. (2003) Blue light- and phosphorylation-dependent binding of a 14-3-3 protein to phototropins in stomtal guard cells of broad bean. Plant Physiol. 133: 1453-1463
- Kinoshita T, Doi M, Suetsugu N, Kagawa T, Wada M, Shimazaki K. (2001) phot1 and phot2 mediate blue light regulation of stomatal opening. Nature 414: 656-660.
- Inoue S, Kinoshita T, Matsumoto M, Nakayama K, Doi M, Shimazaki K. (2008) Blue light-induced autophoshporylation of phototropin is a primary step for signaling. Proc. Natl. Acad. Sci. USA 105, 5626-5631.
- Takemiya A, Inoue S, Doi M, Kinoshita T, Shimazaki K. (2005) Phototropins promote plant growth in response to blue light in low light environments. Plant Cell 17: 1120-1127.
- Li J, Kinoshita T, Pandey S, Ng CKY, Gygi ST, Shimazaki K, Assmann AM. (2002) Modulation of an RNA-binding protein by abscisic-acid-activated protein kinase. Nature 418: 793-797.
- Kinoshita T, Nishimura M, Shimazaki K. (1995) Cytosolic concentration of Ca2+ regulates the plasma membrane H+-ATPase in guard cells of Fava bean. Plant Cell 7: 1333-1342.
- Kinoshita T, Cano-Delgado A, Seto H, Hiranuma S, Fujioka S, Yoshida S, Chory J. (2005) Binding of brassinosteroids to the extracellular domain of plant receptor kinase BRI1. Nature 433: 167-171.
- Hayashi, Nakamura S, Takemiya A, Takahashi Y, Shimazaki K, Kinoshita T. (2010) Biochemical characterization of in vitro phosphorylation and dephosphorylation of the plasma membrane H+-ATPase. Plant Cell Physiol. 51, 1186-1196.
研究内容(中道)
地球上の生物は、地球の自転に伴う昼夜変動に適応している。概日リズムはその適応に関わる約24時間周期の生物リズムで、概日リズムは「概日時計」と言われる遺伝的に組み込まれた機構によってうみだされている(参考文献1)。概日リズムは多くの生物 (光合成細菌、真菌、植物、昆虫、動物)に見られるが、それらの種に備わっている概日時計は進化的に異なる遺伝子群によって出来上がっていることが近年に研究により明らかとなってきた。 植物の概日時計は、20世紀前半にその存在が示唆されていたが、その実体は長らく謎であった。我々はシロイヌナズナ培養細胞での概日リズムの検出に成功し、概日時計が細胞内で備わっている事を示唆した (2、3)。
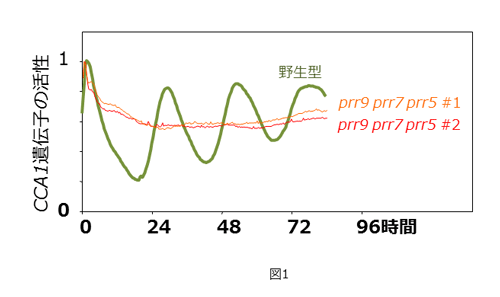
一方、概日時計が1990年以降の研究により、概日時計に関わる遺伝子が続々と発見されてきた。シロイヌナズナのPSEUDO-RESPONSE REGULATOR (PRR)遺伝子ファミリーは、PRR9、PRR7、PRR5、PRR3、PRR1(TIMING OF CAB EXPRESSION 1 [TOC1]と同一遺伝子)は名古屋大学農学部の水野猛教授らが、「遺伝子発現が概日リズムを刻むもの」として2000年に報告している(4)。私たちは、この遺伝子群の解析を進めPRR9、PRR7、PRR5遺伝子のいずれかの機能を欠損すると概日リズムの周期が変わることを示した(5)。PRR遺伝子群は発現時間がオーバラップしており、タンパク質配列も極めて似ているため、機能の重複性が考えられた。そこでさらにPRR9 PRR7 PRR5遺伝子の機能を全て欠損させた植物体を作成し、この植物を解析した(6、7、8)。この植物では 観察した全てのリズムマーカー遺伝子に関して概日リズムが消失していたことから、PRR9、PRR7、PRR5は冗長的に概日時計に必須な因子であることが分かった(図1)。
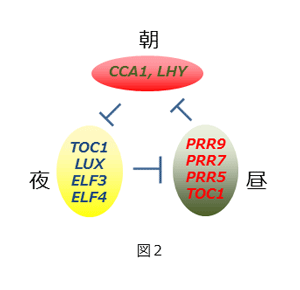
遺伝子の情報配列を眺めても、概日時計の仕組みは理解できない。遺伝子情報が、転写•翻訳され出来上がるタンパク質(生命現象の実行因子)の性質を記述することが時計の仕組みを知る一歩だ。PRRタンパク質の生化学的な機能は分かっていなかったが、我々はその機能を突き止めることにも成功した(9)。まずPRR9、PRR7、PRR5タンパク質に転写抑制活性があることを実験的に突き止め、さらに生体内においてCCA1、LHYという時計関連遺伝子への転写抑制因子として振る舞うことを明らかにした。またPRR9は午前中、PRR7は昼前から夜半、PRR5は夕方から夜半までの「特有の時間」にCCA1およびLHYの遺伝子発現を抑制していることが分かった。最近の世界的な研究結果より朝方に機能する遺伝子CCA1とLHY、昼から夜半に働くPRR9、PRR7、PRR5、TOC1、さらに夜間に働くTOC1、LUX、ELF3、ELF4らが転写フィードバックを形成していることが示されている(10、図2)。
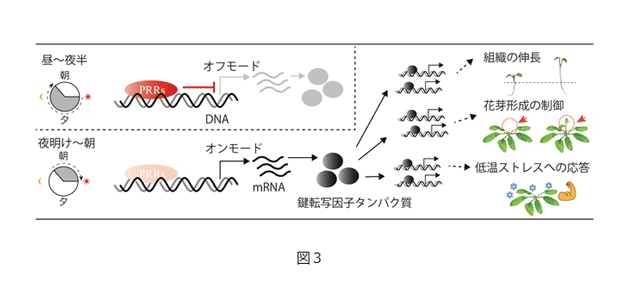
古くから植物における重要な生理現象(花芽の形成、組織の伸長、低温ストレスへの応答)は一日のうちの特定の時間帯にのみ認められることが知られていたが、これらの生理現象の発現時間を決める仕組みは分かっていなかった。我々は転写調節因子であるPRR5が生体内で結合するゲノムDNA配列を、高速シークエンサーで決定する事で、PRR5の直接的なターゲット遺伝子を同定した (11)。これらの遺伝子の多くは、PRR5だけでなく、PRR7とPRR9にもターゲットされていた。60個のターゲット遺伝子の発現の抑制のタイミングは、昼から夜半に認められた。この時間帯は3つのPRRが機能している時間と同じであったため、PRRはこれら遺伝子の発現のタイミングを決める上で主要な抑制因子として振舞うことが示唆された。PRRのターゲット遺伝子には、花成の時期の決定、組織の伸長、そして低温ストレスへの応答の鍵となる転写因子タンパク質をコードしているものがあり、概日時計からこれら生理現象への制御経路が具体的に明らかとなった(図3)。
参考文献(中道)
- 田澤仁「マメから生まれた生物時計: エルヴィンビュニングの物語」学会出版センター
- Nakamichi N, Ito S, Oyama T, Yamashino T, Kondo T, Mizuno T. (2004) Characterization of plant circadian rhythms by employing Arabidopsis cultured cells with bioluminescence reporters. Plant Cell Physiol. 45:57-67.
- Nakamichi N, Matsushika A, Yamahisno T, Mizuno T. (2003) Cell autonomous circadian waves of the APRR1/TOC1 quintet in an established cell line of Arabidopsis thaliana. Plant Cell Physiol. 44:360-365.
- Matsushika A, Makino S, Kojima M, Mizuno T. (2000) Circadian waves of expression of the APRR1/TOC1 family of Pseudo-response regulators in Arabidopsis thaliana: Insight into the plant circadian clock. Plant Cell Physiol. 41:1002-1012.
- Yamamoto Y, Sato E, Shimizu T, Nakamichi N, Sato S, Kato T, Tabata S, Nagatani A, Yamashino T, Mizuno T. (2003) Comparative genetic studies on the APRR5 and APRR7 genes belonging to the APRR1/TOC1 quintet implicated in circadian rhythm, control of flowering, and early photomorphogenesis. Plant Cell Physiol. 44:1119-1130.
- Nakamichi N, Kita M, Ito S, Yamashino T, Mizuno T. (2005) Three Pseudo-response regulators, PRR9, PRR7, and PRR5 together play an essential role close to the circadian clock in Arabidopsis thaliana. Plant Cell Physiol. 46:686-698.
- Nakamichi N, Kusano M, Fukushima A, Kita M, Ito S, Yamashino T, Saito K, Sakakibara H, Mizuno T. (2009) Transcript profiling of an Arabidopsis PSEUDO RESPONSE REGULATOR arrhythmic triple mutant reveals a role for the circadian clock in clod stress responses. Plant Cell Physiol. 50:447-462.
- Fukushima A, Kusano M, Nakamichi N, Kobayashi M, Hayashi N, Sakakibra H, Mizuno T, Saito K. (2009) Impact of clock-associated Arabidopsis pseudo-respoonse regulators in metabolic coordination. PNAS 106:7251-7256. Evaluated in Faculty of 1000 Biology.
- Nakamichi N, Kiba T, Henriques R, Mizuno T, Chua NH, Sakakibara H. (2010) PSEUDO-RESPONSE REGULATOR9, 7, and 5 are transcriptional repressors in the Arabidopsis circadian clock. Plant Cell. 22:594-605. Evaluated in Faculty of 1000 Biology.
- Nakamichi N. (2011) Molecular mechanism underlying the Arabidopsis circadian clock. Plant Cell Physiol. 52:1709-1718.
- Nakamichi N, Kiba T, Kamioka M, Suzuki T, Yamashino T, Higashiyama T, Sakakibara H, Mizuno T. (2012) Transcriptional repressor PRR5 directly regulates clock-output pathways. PNAS, 109:17123-17128.